KaggleのTitanicにトライしてみた話
右も左も分からない人間が、kaggleに挑戦してみた話です。
とりあえず登録してこのページに行ってtrain.csv, test.csvをダウンロード。
train.csvで予測モデルを構築して、test.csvを予想して提出という形。
とりあえず色々データを覗いてみる。
import pandas as pd import matplotlib.pyplot as plt %matplotlib inline train=pd.read_csv("./Titanic/train.csv") train.head(20)
こんな感じに先頭の20個を表示してみると、Cabinに欠損値が多いことが分かる。Name、Sex、Ticket、Cabin、Embarkedは数値じゃないので、捨てるか数値に変換したい。
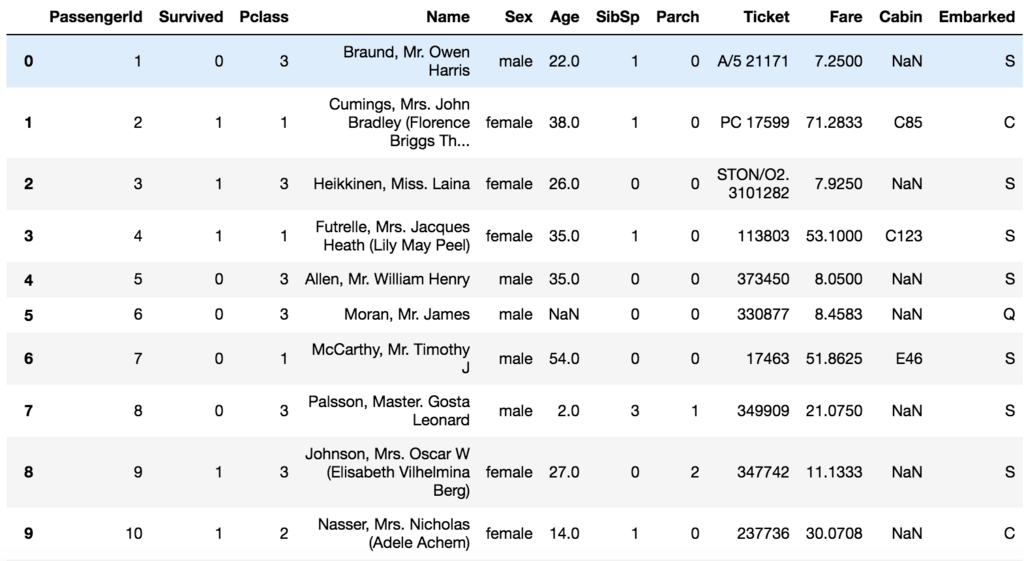
ヒストグラムにしてもう少し覗いてみる前に、SexとEmbarkedを数値に変換して、Name、Ticket、Cabinのデータは捨てることにする。ヒストグラムにする際には、欠損値はdropna()で落としておく。
train.replace({"male": 0, "female": 1}, inplace=True);
train.replace({"Q": 0, "S": 1, "C": 2}, inplace=True);
train.drop(["Name", "Ticket", "Cabin"], axis=1, inplace=True);
dead_data=train[train.Survived==0]
survived_data=train[train.Survived==1]
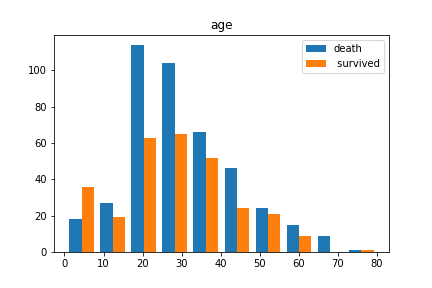
plt.hist([dead_data.Age.dropna(), survived_data.Age.dropna()], label=["dead"," survived"])
plt.legend()
plt.titile("age")
plt.show()
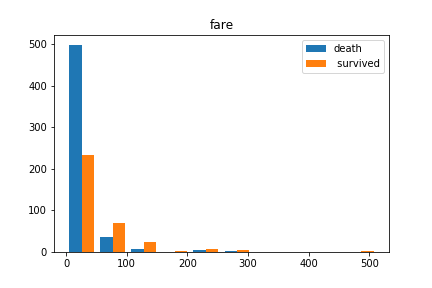
plt.hist([dead_data.Fare.dropna(), survived_data.Fare.dropna()], label=["dead"," survived"])
plt.legend()
plt.titile("fare")
plt.show()
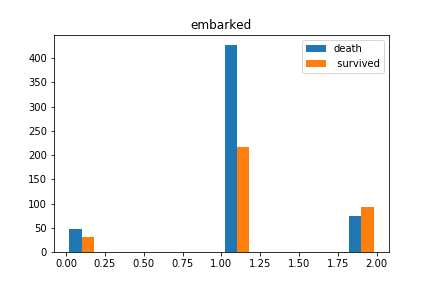
plt.hist([dead_data.Embarked.dropna(), survived_data.Embarked.dropna()], label=["dead","survived"])
plt.legend()
plt.titile("embarked")
plt.show()
欠損値の補完は適当に中央値にしておく。
train.Age.fillna(test.Age.median(), inplace=True); train.Fare.fillna(test.Fare.median(), inplace=True); train.Embarked.fillna(1, inplace=True);
今回行う学習は2値分類なので、有名どころのSVMとRandomforestの比較をしてみる。
データをtrain/testに分けたいときは、sklearnのtran_test_splitが便利。
分けたあとは、ラベルと入力を分けて学習させて評価すれば終わり。
import numpy as np from sklearn.cross_validation import train_test_split from sklearn.ensemble import RandomForestClassifier from sklearn import svm from sklearn.metrics import classification_report, accuracy_score train1, test1=train_test_split(train, test_size=0.2, random_state=None) train_feature=train1.drop("Survived", axis=1) train_label=train1.Survived test_feature=test1.drop("Survived", axis=1) test_label=test1.Survived clf = svm.SVC() clf.fit(train_feature, train_label) svm_pred=clf.predict(test_feature) print(classification_report(test_label, svm_pred)) print(accuracy_score(test_label, svm_pred)) rf=RandomForestClassifier() rf.fit(train_feature, train_label) rf_pred=rf.predict(test_feature) print(classification_report(test_label, rf_pred)) print(accuracy_score(test_label, rf_pred))
こちらの手元では、SVMが0.6、RFが0.8弱だったので、RFを採用して提出することにした。
#testデータの前処理 test.replace({"male": 0, "female": 1}, inplace=True); test.replace({"Q": 0, "S": 1, "C": 2}, inplace=True); test.drop(["Name", "Ticket", "Cabin"], axis=1, inplace=True); test.Age.fillna(test.Age.median(), inplace=True); test.Fare.fillna(test.Fare.median(), inplace=True); test.Embarked.fillna(1, inplace=True); rf=RandomForestClassifier() rf.fit(train.drop("Survived", axis=1), train.Survived) rf_pred_all=rf.predict(test) submission = pd.DataFrame() submission['PassengerId'] = test.PassengerId submission['Survived'] = rf_pred_all submission.to_csv('submission.csv', index=False)
が、、、ここまで来てKaggleのページからどうやってsubmitすればいいのか分からなくなった。。。
調べたところ、最近になってKaggleはAPIで提出する形になったようなので仕方なくpip install kaggle。kaggleのページに行って、"My account">"create new api token"に進むと、kaggle.jsonのダウンロードが開始される。homeディレクトリに.kaggleを作成してkaggle.jsonをそこに入れたのち、
!kaggle competitions submit -c titanic -f submission.csv -m "RF all data"
で提出完了!データを落としてくるところもAPIでいけるらしい。ほー。
感想とか。
Embarkを0,1,2にしちゃダメそうな気がしたけど、まあとりあえずやっちゃった感ある。
SVMの精度がめちゃめちゃ悪かったのがなんでなのかよく分からない。ここでは結構精度が出てるので、どこかが変なのかもしれない。
前処理はもっと工夫するべきなんだろうけど、初めてのKaggleということで大目に見てもらいたい。
コネクトームをシーケンサーに掛けたい
今年読んだ一番好きな論文2017の12月21日の記事です。
初めまして。kira_kira_worldです。合成生物学と基礎医学が好きな学部生です。この企画は以前から見ていて、ずっと見る専だったんですが、今年は景品欲しさに登録してみました。
紹介論文
紹介する論文は、"Using high-throughput barcode sequencing to efficiently map connectomes"です。バーコード技術(後述)を使ってコネクトームを決定する方法を考えましたっていう論文です。
前置き
Wikipediaの"コネクトーム"というページを引用しますと、
「コネクトーム(connectome)とは、生物の神経系内の各要素(ニューロン、ニューロン群、領野など)の間の詳細な接続状態を表した地図、つまり神経回路の地図のこと。」
とのことです。今回の論文の中では、コネクトームとは神経細胞の全接続のことを指しますが、コネクトームの全体像を描くのはかなり難しいようで、線虫(めっちゃ小さい生き物)とかでしかコネクトームは分かっていないようです。現状、コネクトームを網羅的に決定するには顕微鏡を用いるしかありませんが、脳全体を顕微鏡で少しずつ見ていくことはほぼ不可能なので、コネクトーム決定の困難さはこのことからもわかります。
そこで、いい感じにハイスループットにポンポンポン!ってなんねぇかな〜〜ってことで、著者たちがいい感じの方法を思いついたみたいです。ワクワクしますね、一体どんな方法なんでしょう。
基本的なアイデア
アイデアとしては、シナプス前膜と後膜それぞれを異なる配列の短いRNA(バーコード)で以って標識してやり、なんとかして一つの配列に繋げてシーケンサーに突っ込めば、各シナプス結合がタンデム配列として認識できるだろうというものです。
具体的には、NeurexinとNeuroliginというシナプス前後に局在して手をつなぐタンパク質を利用します。RNA結合モチーフを細胞質側に融合し、細胞外にはリンカー結合部位を融合します。すると、バーコード配列はシナプスに局在し、ビオチンの付いたリンカーを加えてやればタンパク質は架橋されます。その後、細胞を溶解させ、ビオチンで免疫沈降すれば、RNAのペアが取得できます。名付けてSYNseqと言うらしいです。なんて知的でアクロバティックかつ繊細なアイデアなんでしょう!

実験結果
新しくてかなりアクロバットな手法を開発しようとしているため、細かいvalidationが多かったみたいです。論文に記述しきれていない実験が大量にある雰囲気が読んでてバシバシ伝わってきます。実験内容について簡単に説明すると、著者たちは、
✅ RNAとRNA結合モチーフが結合して細胞膜に局在する(HEK細胞&ニューロンにおいて)
✅ シナプスで両方のタンパクは近傍に存在している
✅ 架橋し、免疫沈降で落ちてくる
という確認をしたようです。(詳細は省略)
続いて、RNA同士を結合させるという部分ですが......すでにここまでの実験でかなり大変だったみたいなのに、ここが最大の壁だったようです。著者たちは、色んな方法を試してみて(splinted ligation, ribozyme)、エマルジョン overlap&RT-PCRという方法に行き着きました。
エマルジョンPCRとは、油中に非常に小さな水滴を作り、その水滴を小さな反応炉として利用することで、沢山の種類の核酸を別個に増幅させる方法です。通常、次世代シークエンサーのサンプル調整で使用されます(例えばこのページを参照)。

ということで、全体のフローがようやく完成しました。とりあえずHEK細胞でワークするか試した結果が下の図です。preまたはpostのタンパク質を発現させた細胞を混ぜたあとにシークエンスしてあります。縦軸・横軸がそれぞれHEK細胞にトランスフェクションしたバーコード配列(即ち個々の細胞)、各点がそれらの接続を意味します。

おお、なんかそれっぽそう!
では、ニューロンでは?というと、HEK細胞での実験と似たような手法を用いて実験を行いましたが、感度が非常に悪くなってしまい、RNAをほとんど検出できなかったようです。図すらありませんでした。これはどうやらRNAの結合効率が悪かったせいのようです。著者らの実験はここで終了しています。*1
(他にも色々書かれているけど省略)
結論
今回の結果ではコネクトームは読めない。
感想とか
手法の正しさをどうやって証明するのか、preとpostのタンパク質は本当にニューロンで正しい局在をするのか (細胞膜全体に局在していそうな図がある)、1細胞内でcross-linkしてしまうのではないか、などと色々と疑問が多い論文でした。
結果は全然ダメでしたが、発想においては今年読んだ論文で一番面白かったです。今回の記事ではかなり省略しましたが、色々な分子生物学的なギミックが本論文では使われており、そういうツールを使って生物学に切り込んでいくのは楽しそうだなと思いました(結果が伴えば)。僕にとっては技術的に難しい部分が多く、非常に勉強になりました。気になった方は読んでみてください!(そして間違いがあったら指摘してください。)
UCSD Roy lab その3
これ。
顕微鏡の自動サンプリングシステムを使って、aSynによるvesicleダイナミクスのunbiasedな解析をした論文。系が面白い。もう写真撮る過程のbiasは無いものと信じるしかないからな。「PDと小胞輸送の関連はもう前から言われていたけど、aSynはpreにいるからER→Golgiの話をするのはおかしい」とのこと。当たり前だけど、文献のアブストの内容くらいしか覚えてないようだとここら辺はあまり意識しないかもな。まあでも最近はdendriteに蛋白合成系のオルガネラは揃ってるみたいな話もあったりするから、もしかしたらpreにも系が全部揃ってる可能性はある気がする。オートファジーとかなら、どこのオルガネラがオートファジーを受けるのかってことになってくるのかな。
やっぱりこの論文の1stの方、この分野続けて欲しかったなぁ。
メモ
エクソソームについて調べていたところ、まとまった日本語のページがあったので貼っておく。
UCSDのRoy labの続き
先日のRoy labから出た論文が非常に参考になると分かったので、しばらくはここの研究室の論文を読むことにする。
とりあえずこれ。アルツハイマー病(AD)はAPPがγ, β secretaseによって切断されることによって生成されたAβが凝集することが特徴的。最も多い神経変性疾患。
APPがendocytosisされて、BACE-1のいるRecycling endosomeに取り込まれて処理を受けることを報告した論文。ADの人の脳で、実際にAPPとBACE-1の分画が増えていることを最後に示している。activity dependentらしく、デフォルトネットワークの活動度がADと関与しているのではないかとの報告もあるようで、その具体的なメカニズムではないか?と言っている。ほとんどイメージングしかしてない。
なお、ニューロンは全て初代培養で、lentiか何かでADのモデルにしたものは使ってない。実際にADのGWASか何かで出てきたSNPはこの経路に関するものなのだろうか?sporadicなADの関与とも関係ありそうだ。